| LIAISON AVEC LE PROGRAMME | |
|---|---|
| Niveau concerné | Spécialité de Première |
| Partie du programme : | La Terre, la vie et l’organisation du vivant - L’histoire humaine lue dans son génome |
| PLACE DANS LA PROGRESSION |
|---|
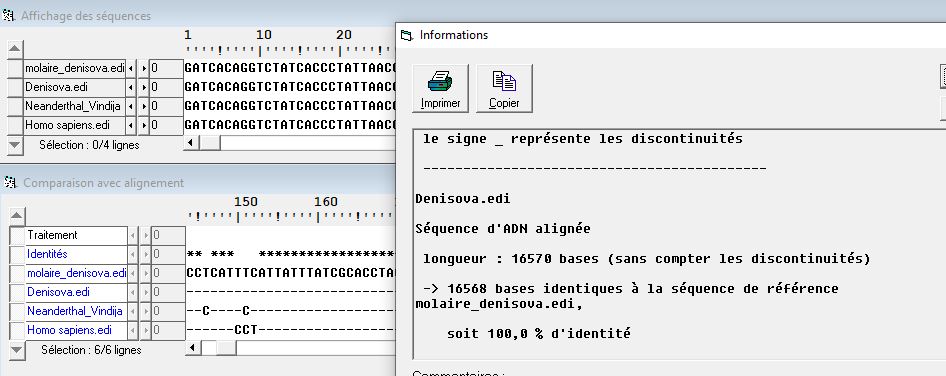
| Les activités proposées interviennent après que la partie Mutations de l’ADN et variabilité génétique ait été traitée. L’origine des changements de séquence de l’ADN est donc connue. Les comparaisons de séquence dans Anagène ont été utilisées pendant ces parties précédentes |
| NOTIONS,COMPÉTENCES/CAPACITÉS | |
|---|---|
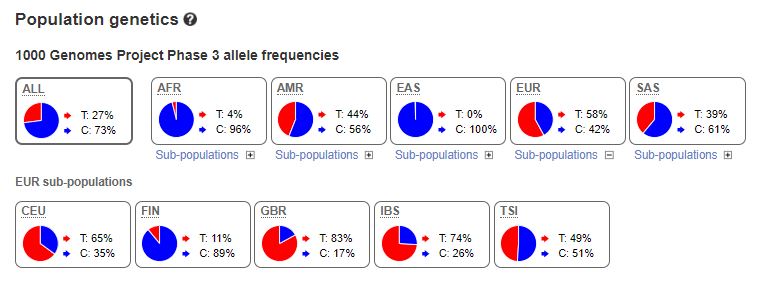
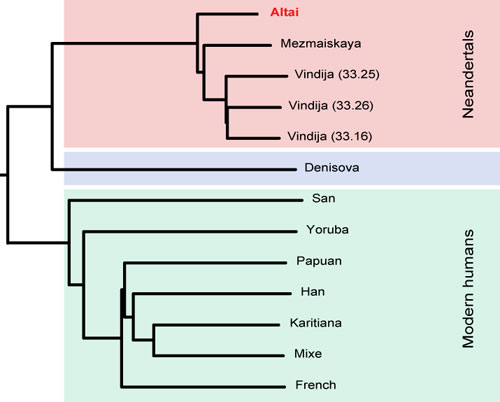
| Notions | La diversité allélique entre les génomes humains individuels permet de les identifier et, par comparaison, de reconstituer leurs relations de parentés. Grâce aux techniques modernes, on peut connaître les génomes d’êtres humains disparus à partir de restes fossiles. En les comparant aux génomes actuels, on peut ainsi reconstituer les principales étapes de l’histoire humaine récente. Certaines variations génétiques résultent d’une sélection actuelle (tolérance au lactose, résistance à la haute altitude) ou passée (résistance à la peste). |
| Compétences/Capacités | Explorer quelques stratégies et outils informatiques de comparaisons de séquences entre génomes individuels. Rechercher et exploiter des documents sur les génomes de néandertaliens et/ou de denisoviens. Rechercher et exploiter des documents montrant l’existence d’allèles néandertaliens dans les génomes humains actuels |
| ACTIVITE | ||
|---|---|---|
| Activité 1 : qui sont les Denisoviens ? | Activité 2 : Retrouver ses origines grâce à l’ADN ? | |
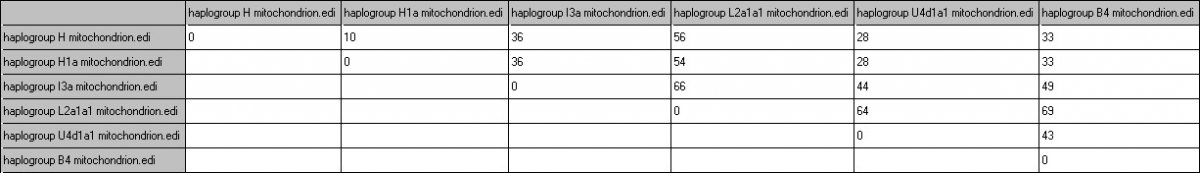
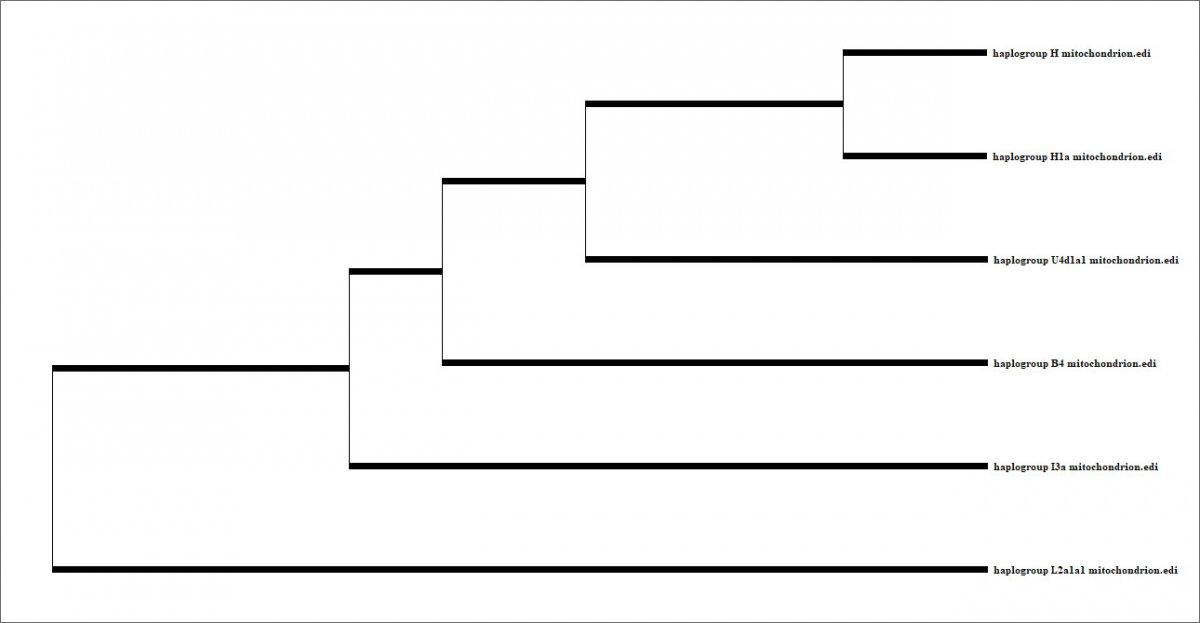
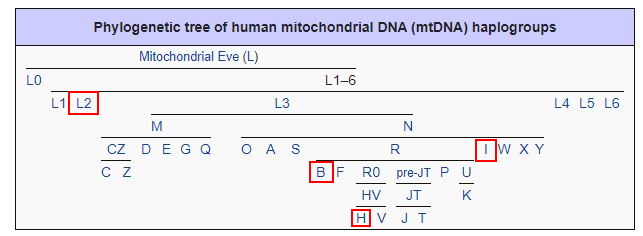
| Activité 3 : Une histoire des populations d’après l’étude des haplogroupes | ||
| Activité 4 : L’origine de l’adaptation à l’altitude des Tibétains sur la plateforme ACCES | Un autre exemple : La persistance à la lactase sur la plateforme ACCES | |
| L’activité 4 n’est pas une proposition originale. On a cherché ici à l’articuler dans une démarche d’ensemble. L’activité 2, si elle peut sembler hors programme, permet à la fois d’introduire les SNP qui seront nécessaires pour l’activité 4 et d’aborder un phénomène de mode actuel qui pose question. Ce ne sont que des propositions d’activité dans une proposition de séquence. On pourra choisir de réaliser tout ou partie de ce qui est proposé, ou articuler autrement ces activités ou les articuler avec d’autres, sur d’autres exemples. | ||
| Durée : 4h | Coût : 0 euros | Sécurité : RAS |
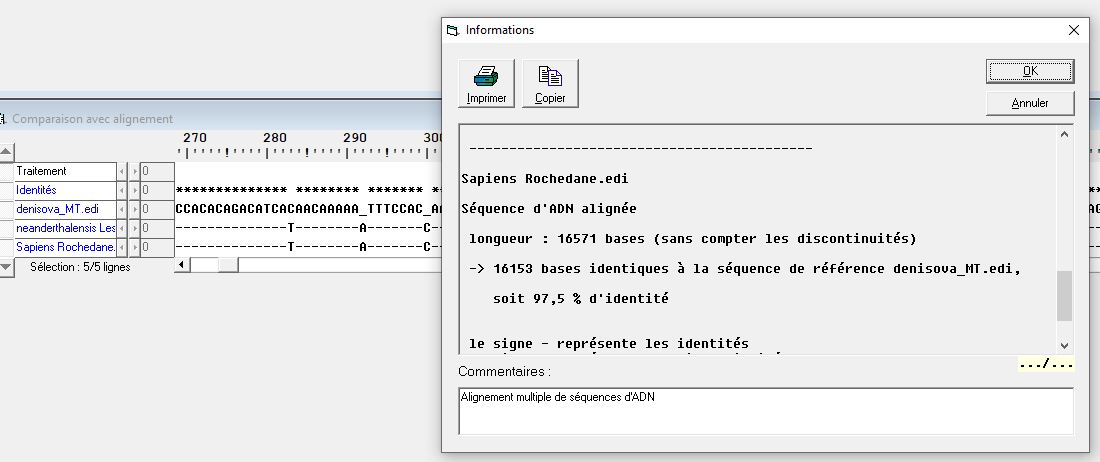
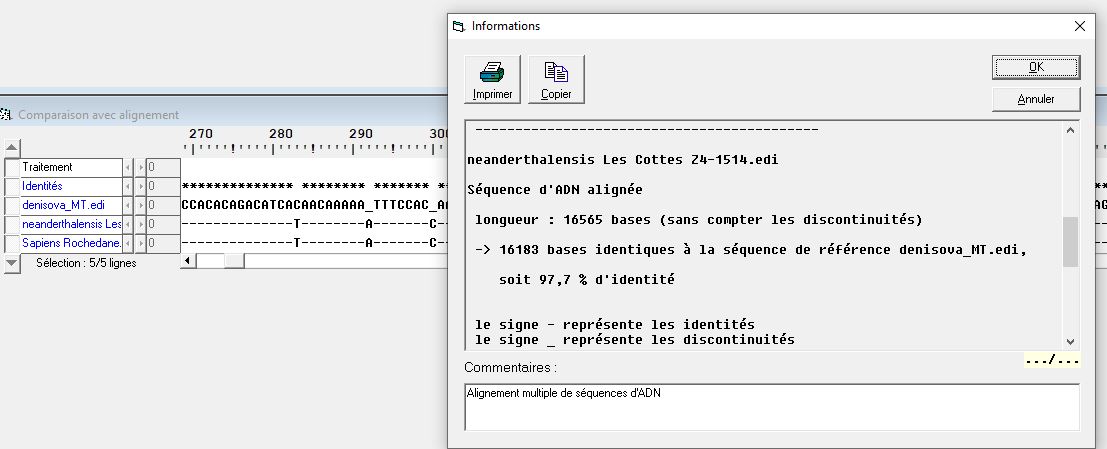
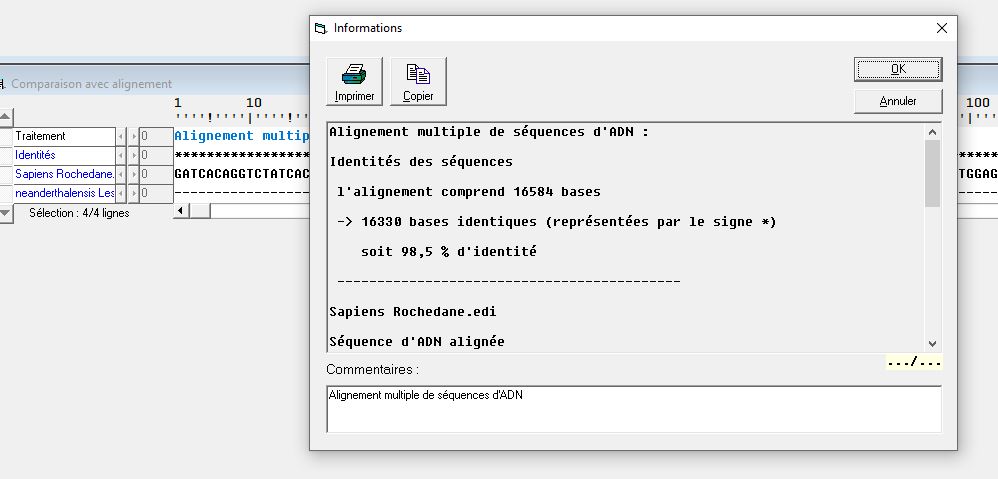
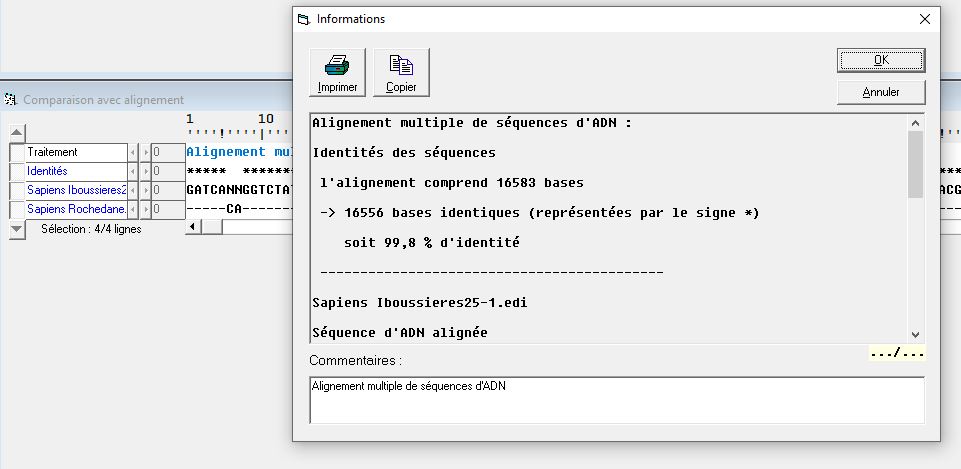
| Matériel et ressources : ordinateurs, accès à internet, logiciel Anagène ou autre logiciel permettant la comparaison de séquences, logiciel Phylogène. Pour ces propositions, je me suis efforcé d’utiliser ces outils qui nous sont familiers. Il faut néanmoins avoir conscience qu’ils ne sont pas bien adaptés à cette nouvelle partie de programme. Les séquences à utiliser sont trop lourdes pour ces logiciels : c’est pourquoi les activités proposées restent beaucoup sur l’ADN mitochondrial qui est petit comparativement aux chromosomes nucléaires qu’Anagène ne peut traiter. (Nous sommes dans la même situation que les chercheurs au début de ces études de paléogénomique). Même pour l’ADN mitochondrial, on verra que les temps de calcul sont assez longs. Pour Phylogène, lors de la préparation du fichier de séquence, un message indiquait que l’alignement des séquences n’était que partiel. Mais l’inadaptation des logiciels est surtout liée au fait que l’analyse des parentés et origines se fonde plutôt sur l’étude de SNP spécifiques, des variations ponctuelles, et non sur une comparaison globale des séquences avec des % de ressemblance. | ||