Libmol.org est un logiciel de visualisation moléculaire libre et gratuit, à la prise en main aisée pour les élèves, créé par Paul Pillot. Il en existe une version en ligne et une autre téléchargeable. Ses fonctionnalités sont décrites dans cet article : Libmol.org, nouvelle application de visualisation de molécules, alternative à RasTop.
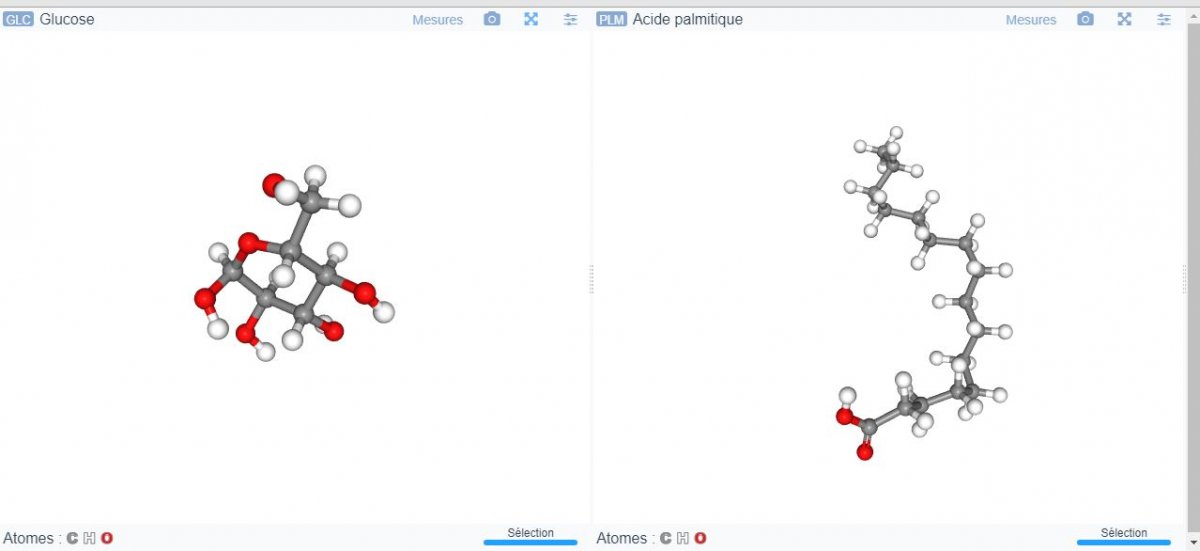
Cependant, une fonctionnalité de RasTop intéressante pédagogiquement, la visualisation « en cascade » ou en fenêtres multiples, n’a pas été développée pour l’instant. Il est pourtant important pour les élèves de pouvoir comparer les structures moléculaires en les plaçant côte à côte, par exemple en Seconde pour distinguer les éléments chimiques du vivant de ceux du monde minéral, ou encore en Première S pour comprendre la structure des acides aminés.
Pour pallier à ce manque, plusieurs possibilités s’offrent à l’enseignant avec la version en ligne, au delà de simplement proposer aux élèves d’ouvrir plusieurs fenêtres du navigateur, y taper à chaque fois l’adresse du site, redimensionner et placer les fenêtres les unes à côté des autres.
Utiliser une visionneuse en ligne intégrant plusieurs fenêtres Libmol
Nous vous proposons cette interface utilisable en ligne, permettant d’afficher au choix 1 à 4 molécules dans la même page.
Attention : L’interface fonctionne de manière optimale avec Mozilla Firefox.
Lien d’accès : https://view.genial.ly/5a05bb3421cf3624787045d3
Voici un lien court et un QR-code à fournir pour utilisation sur tablette :
http://opn.to/a/8ziaa

L’outil est intégrable dans la plupart des ENT ou dans une plate-forme Moodle comme Éléa, grâce à cette ligne de code :
<iframe frameborder="0" width="1200pxpx" height="675pxpx" style="position: absolute; top: 0; left: 0; width: 100%; height: 100%;" src="https://view.genial.ly/5a05bb3421cf3624787045d3" type="text/html" allowscriptaccess="always" allowfullscreen="true" scrolling="yes" allownetworking="all"></iframe>
Utiliser une interface web
Cette interface a été proposée par Philippe Cosentino, de l’académie de Nice, pour comparer deux molécules.
- Lien d’accès : http://philippe.cosentino.free.fr/dual_libmol/
- Lien intégrant directement deux molécules à comparer : exemple pour comparer l’alanine et la glycine, copier-coller cette adresse : http://philippe.cosentino.free.fr/dual_libmol/index.htm?pdb1=ALA&pdb2=GLY
Pour comparer deux autres molécules, il faudra rechercher le code de ces molécules et modifier une partie de l’adresse : pdb1=(suivi du code de la 1ère molécule)& pdb2=(suivi du code la 2ème molécule).
Où trouver ces codes ?
- Il y a d’un côté la banque de macromolécules avec des codes à 4 caractères, par exemple en fouillant dans la Protein Data Bank.
- Il y a aussi le « Chemical component dictionary » qui donne accès à tous les résidus et petites molécules existant dans les fichiers PDB. Ce sont les mêmes identifiants que ceux que l’on peut utiliser pour sélectionner un résidu : ALA, GLY, DA, A, HEM, etc… Pour les rechercher, on peut passer par cette page et choisir l’onglet « Name/Identifier ».
- Vous trouverez aussi directement ces codes dans Libmol, en haut à gauche de la fenêtre quand s’affiche votre molécule.
Merci à Paul Pillot pour ces précisions !