
Comment comparer ou visualiser les structures 3D de molécules ?

On pourra utiliser le logiciel Rastop

 Ouverture d'un fichier
Ouverture d'un fichier
Utiliser la commande 1, ouvrir le fichier demandé en suivant le chemin d’accès.
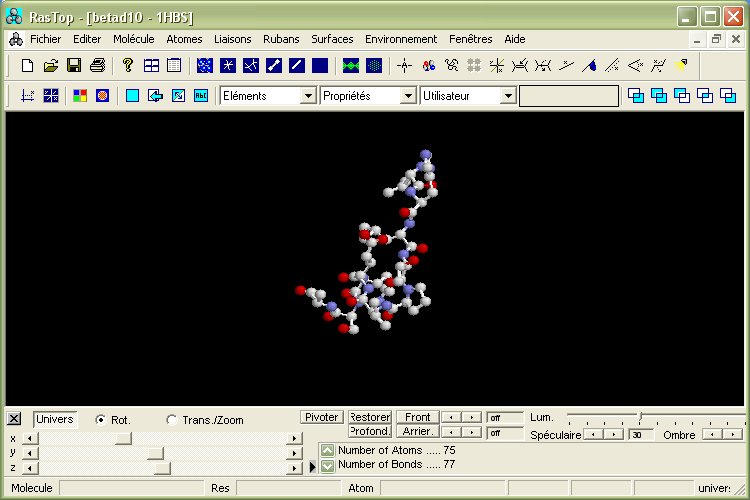
 Visualisation d'une molécule
Visualisation d'une molécule
![]() Choix d’un mode de représentation : utiliser au choix l’une des commandes 2 .
Choix d’un mode de représentation : utiliser au choix l’une des commandes 2 .
Les atomes sont colorés de manière conventionnelle : gris clair = Carbone(C) ; rouge = Oxygène (O); bleu clair = Azote (N) et orange = Phosphore (P) [H l’hydrogène n’est pas représentée].
![]() Manipulation de l’image de la molécule :
Manipulation de l’image de la molécule :
cliquer dans l’écran noir avec le bouton gauche de la souris et sans relâcher la pression sur ce bouton, déplacer lentement la souris : vers la gauche ou la droite pour tourner autour de l’axe des y, vers le haut ou le bas pour tourner autour de l’axe des x.
![]() Zoom avant ou arrière : soit en cliquant sur le bouton trans./zoom puis en bougeant le curseur des z ; soit avec la souris en maintenant la touche Maj. appuyée.
Zoom avant ou arrière : soit en cliquant sur le bouton trans./zoom puis en bougeant le curseur des z ; soit avec la souris en maintenant la touche Maj. appuyée.
 Mise en évidence de la constitution d'une molécule
Mise en évidence de la constitution d'une molécule
![]() Coloration des constituants : Utiliser la commande 3, puis « colorer par » puis choisir dans la liste.
Coloration des constituants : Utiliser la commande 3, puis « colorer par » puis choisir dans la liste.
![]() Coloration des liaisons : Utiliser la commande 4 puis « liaisons hydrogène » et « afficher » ou « ponts disulfure » et « afficher ».
Coloration des liaisons : Utiliser la commande 4 puis « liaisons hydrogène » et « afficher » ou « ponts disulfure » et « afficher ».
![]() Informations sur la séquence : Chaque élément d’une séquence est symbolisé par une ou plusieurs lettres suivies d’un numéro correspondant à sa place. Il faut lire les informations :
Informations sur la séquence : Chaque élément d’une séquence est symbolisé par une ou plusieurs lettres suivies d’un numéro correspondant à sa place. Il faut lire les informations :
- soit au bas de l’écran dans la zone nommée « res » Dans l’exemple présenté ci-dessous,, l’élément considéré est la Thymine placée en 20° position dans la chaîne B de la molécule 1D29 (ADN)
![]()
- Soit en utilisant la commande 6 puis « séquence »
![]() Mise en évidence d'une partie de la molécule
Mise en évidence d'une partie de la molécule
![]() Sélection du motif qu’on souhaite faire ressortir : utiliser la commande Expression : bouton 6
Sélection du motif qu’on souhaite faire ressortir : utiliser la commande Expression : bouton 6
Taper ce qu’on souhaite mettre en valeur, par exemple :
26-28, ce qui signifie que vous sélectionnez les acides aminés 26 à 28 de la séquence de la chaîne polypeptidique constituant la molécule
hem, ce qui signifie que vous sélectionnez simplement l'hème
à partir de là, tous les traitements demandés ne s’appliqueront qu’aux constituants du motif venant d’être sélectionné
![]() Coloration des atomes sélectionnés : Ouvrir la Palette 5, choisir Atomes dans le menu déroulant de la fenêtre Colorer puis choisir à la souris la couleur demandée
Coloration des atomes sélectionnés : Ouvrir la Palette 5, choisir Atomes dans le menu déroulant de la fenêtre Colorer puis choisir à la souris la couleur demandée
